9月24日,國際學術期刊Genome Biology在線發表了中國科學院上海營養與健康研究所生物醫學大數據中心張國慶研究員與南方科技大學生命科學學院系統生物學系王澤峰講席教授團隊的合作論文“A foundation language model to decipher diverse regulation of RNAs”。該研究構建了一種基于深度神經網絡的預訓練模型,可微調預測pre-mRNA的剪接位點、mRNA的翻譯效率、mRNA的降解率和內部核糖體進入位點(Internal Ribosome Entry Site,IRES)等多個RNA調控相關的下游任務,揭示了RNA中調控元件的序列特征并鑒定新型翻譯調控元件,為理解RNA調控機制和優化RNA的生物醫學應用提供了新工具和新思路。
在真核生物中,RNA轉錄、剪接、翻譯和降解等生物學過程受到順式調控元件、RNA結構和反式作用因子的嚴格調控。解析RNA的多層次調控對于研究基因表達分子機制和設計RNA藥物具有重要意義。然而由于調控的復雜和數據量的不足,目前構建RNA調控的預測模型仍然面臨挑戰。
為了突破上述瓶頸,研究團隊設計并訓練了基于多層transformer編碼器架構的RNA語言模型LAMAR。研究首先下載處理約1500萬條哺乳動物和病毒的基因和轉錄本序列,通過掩碼學習進行無監督預訓練,預先提取RNA的序列特征;之后使用含有標簽的數據集微調模型,實現RNA調控高效預測。
研究測試了LAMAR模型在多個下游任務中的性能。其中,LAMAR模型在mRNA翻譯效率和降解率預測任務中分別取得0.66和0.65的Spearman相關系數指標,相比最優基線模型提升7%和8%。另外,LAMAR模型在剪接位點預測任務中取得0.96的PR-AUC指標,與最優基線模型SpliceAI的性能相當。
研究還使用公開數據集微調模型預測病毒和真核IRES,取得0.985的AUROC指標。研究進一步預測RNA病毒基因組中潛在的新IRES,并在多個細胞系中測試其中305條序列驅動環形RNA翻譯的效率。研究發現序列的預測概率與翻譯活性呈正相關,提示模型模擬篩選新型調控元件的能力。
目前,LAMAR模型已上傳至Github(https://github.com/rnasys/LAMAR),供科研人員預測pre-mRNA的剪接位點、mRNA翻譯效率、降解率和IRES,或使用自己的數據集微調模型。
中國科學院上海營養與健康研究所張國慶研究員、南方科技大學生命科學學院王澤峰教授、美國北卡羅萊納大學教堂山分校胡玥博士后為論文共同通訊作者。中國科學院上海營養與健康研究所博士研究生周翰文、美國北卡羅萊納大學教堂山分校胡玥博士后為論文共同第一作者。該研究得到了科技部國家重點研發計劃、國家自然科學基金、中國科學院戰略性先導科技專項(B類)、上海市科技創新行動計劃、上海市市級科技重大專項等項目的資助。
論文鏈接:https://genomebiology.biomedcentral.com/articles/10.1186/s13059-025-03752-x
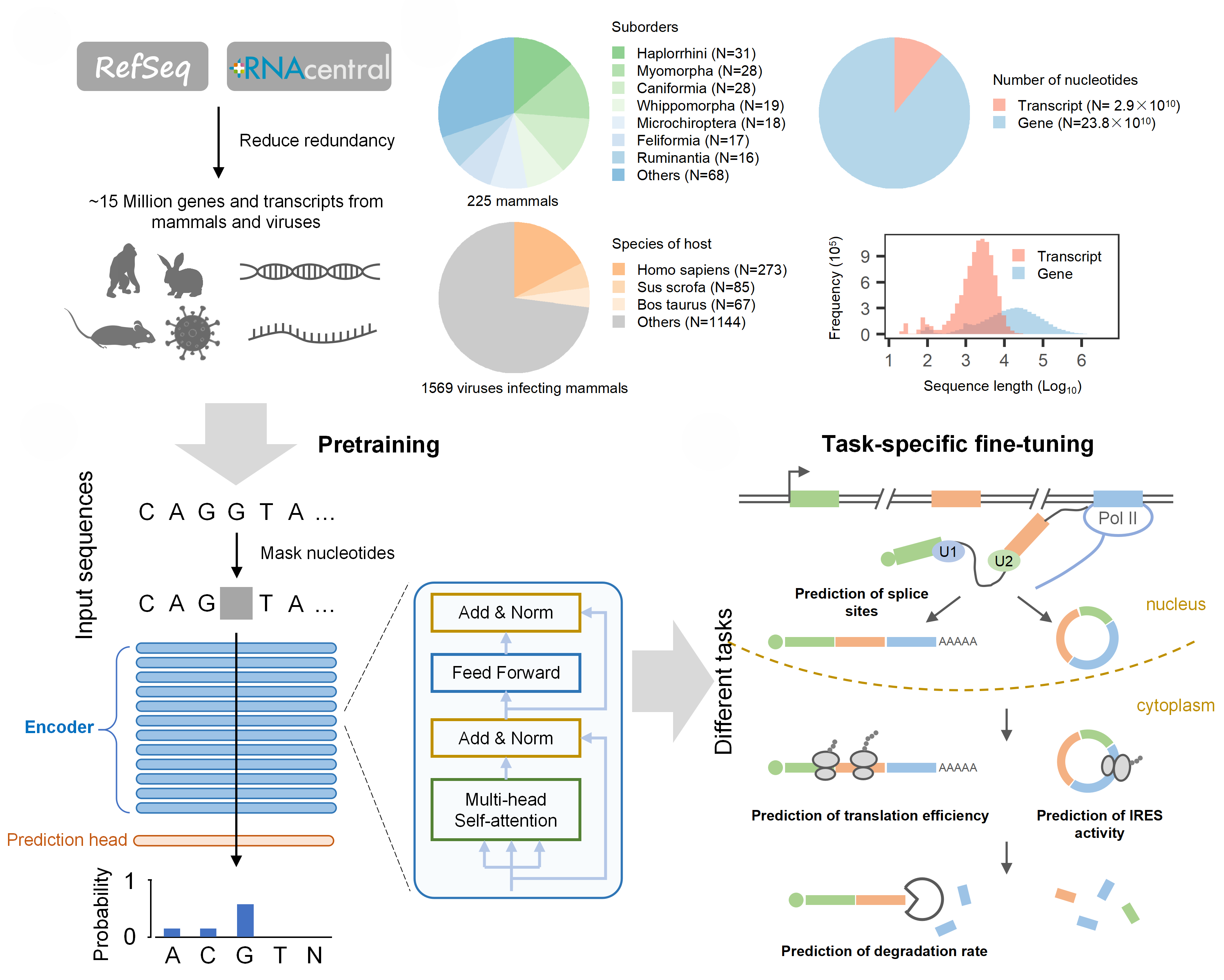
圖1:LAMAR模型架構及研究流程圖:模型首先使用大量基因和轉錄本序列進行無監督預訓練,再使用含有標簽的數據集進行微調解決RNA調控下游任務。
推送單元:生物醫學大數據中心、科技規劃與任務處